Neural Cellular Automata Manifold
原文链接:https://arxiv.org/abs/2006.12155
作者:Alejandro Hernandez Ruiz, Armand Vilalta, Francesc Moreno-Noguer (IRI UPC, BSC UPC)
日期:2021-03
被引用:12(来自谷歌学术)
概要:如果说 Growing CA 是“为一个图案训练一个专门的生物”,那么本文就是“训练一个上帝(元模型)”,它能根据输入的 DNA(潜变量)瞬间制造出能生长成对应图案的生物规则。本文提出 Neural Cellular Automata Manifold (NCAM),通过 Auto-Encoder 和 HyperNetwork 技术,训练一个单一模型来编码和生成成千上万种不同的图案生成规则。
引言
多细胞生物的繁殖涉及从单个细胞生成整个躯体。这一过程依赖于三个支柱:细胞分化、形态发生和细胞生长控制。虽然 Neural Cellular Automata (NCA) 成功模拟了这一过程,但原始 NCA 存在一个主要限制:它是一个“专才”。每个训练好的 NCA 网络只能生成一个特定的图案(例如一只壁虎)。如果想要生成另一个图案(例如一个 Emoji),就需要从头训练一个新的网络。
本文旨在解决这个问题,通过引入“元学习”的思想,训练一个通用的流形 (Manifold) 模型。这个模型可以: 1. 存储效率:用一个网络编码成千上万种图案的生成规则。 2. 泛化能力:学习图案的通用底层结构,允许在潜空间进行插值(混合物种)和生成未见过的变体。 3. 生物学合理性:模拟基因(DNA)如何通过转录因子(Parameter Predictor)调控细胞机器(NCA)的行为。
方法
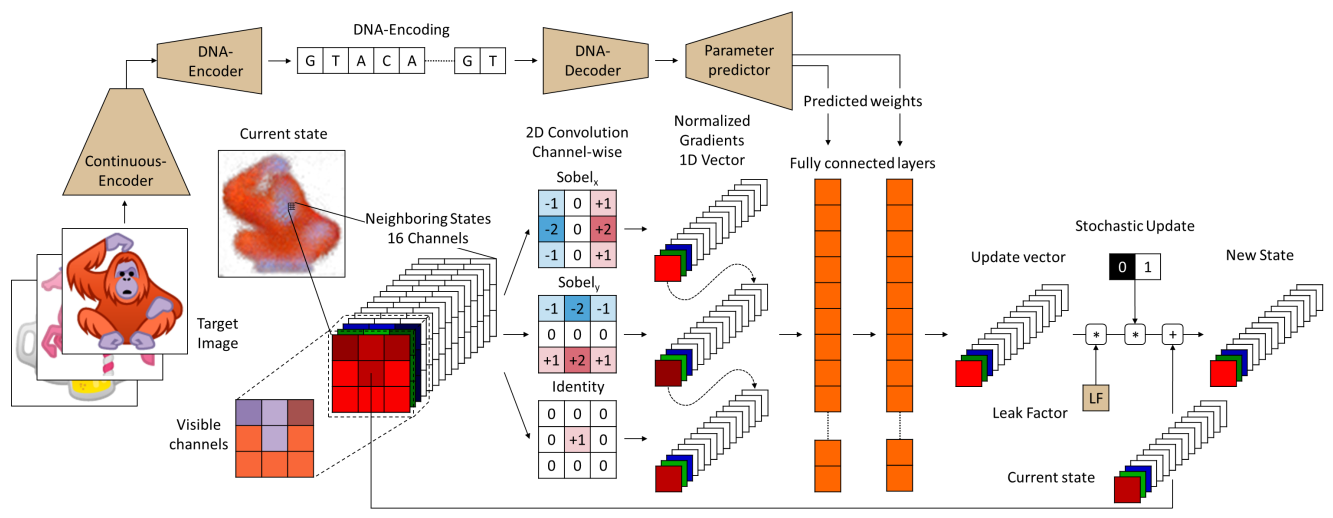
NCAM 将图像生成的形态发生过程建模为一个由输入图像编码控制的动态系统。
状态空间定义
定义时刻 $t$ 的图像状态 $I^t$ 为网格中所有细胞状态的集合。每个细胞 $(i, j)$ 包含: 1. 可见表型 $C_{ij}^t$(RGB/RGBA)。 2. 形态发生素 $M_{ij}^t$(隐藏通道)。
$$ I^t = \{ (C_{ij}^t, M_{ij}^t) \} \quad \forall i, j \in \text{Grid} $$
动态更新方程
传统的 CA 使用固定的规则,而 NCAM 的核心在于规则是输入图像 $I$ 的函数。细胞状态的更新方程为:
$$ \begin{aligned} C_{ij}^{t} &= f(C_{ij}^{t-1}, M_{kl}^{t-1}, \kappa(e^{I}, \theta), \theta_{LF}) \\ M_{ij}^{t} &= g(C_{ij}^{t-1}, M_{kl}^{t-1}, \kappa(e^{I}, \theta), \theta_{LF}) \end{aligned} \quad \forall (k, l) \in \epsilon_{ij} $$
其中 $\kappa(e^I, \theta)$ 是动态权重生成函数,它根据图像编码 $e^I$ 生成当前 NCA 的卷积核权重。
权重生成机制 (HyperNetwork)
NCA 的参数由一个元网络生成:
$$ \kappa(e^{I}, \theta) = \mathcal{P}(\mathcal{D}(e^{I}, \theta_{\mathcal{D}}), \theta_{\mathcal{P}}) $$
- $\mathcal{D}(\cdot)$:DNA 解码器。
- $\mathcal{P}(\cdot)$:参数预测器 (HyperNetwork)。
架构详情
整体架构是一个端到端的 Auto-Encoder,但在解码端引入了 HyperNetwork (Parameter Predictor) 和 NCA。
基础构建模块
为了适应重建任务,作者设计了特殊的残差块,不改变空间分辨率,但通过增加通道数来解耦流形。
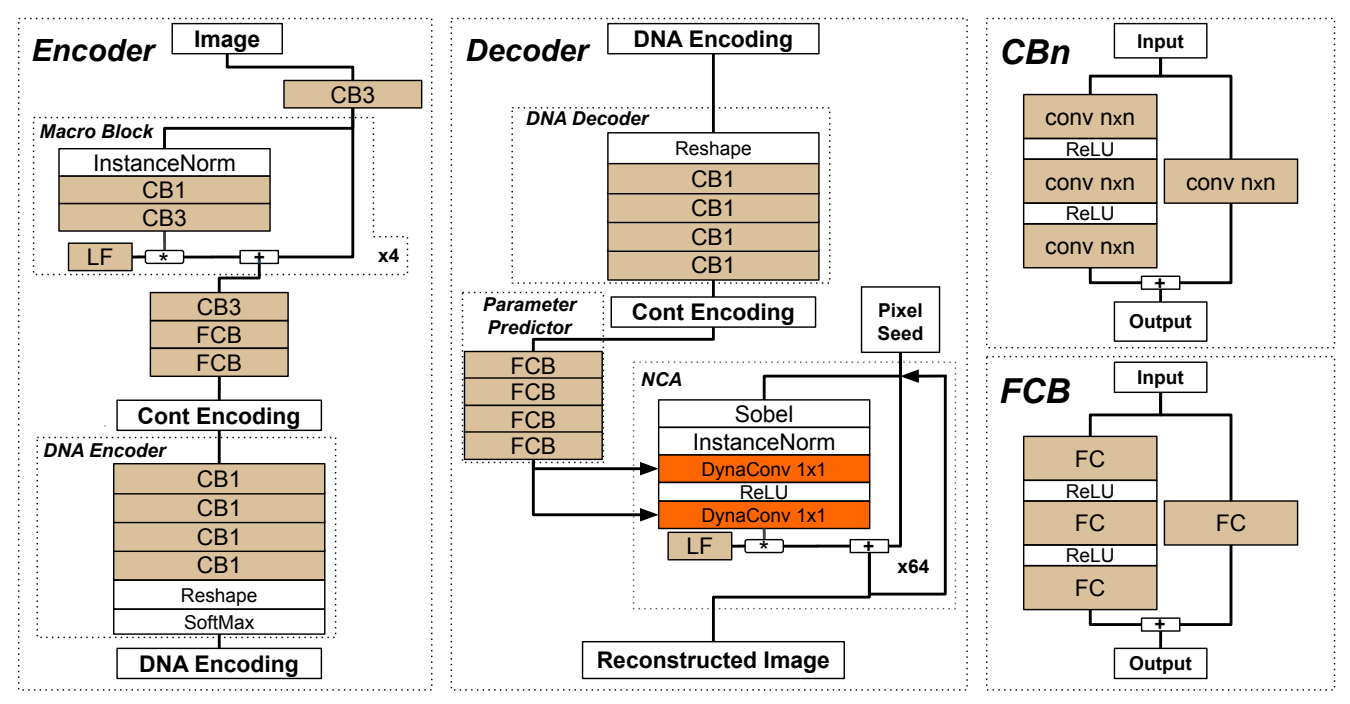
-
CB3 (Convolutional Block 3x3):
- 结构:包含两个 3x3 卷积层的残差块。
- 特征扩展:内部层(Inner Layer)的通道数扩展为输入的 2倍。
- 作用:引入邻域空间信息处理。
-
CB1 (Convolutional Block 1x1):
- 结构:包含两个 1x1 卷积层的残差块。
- 特征扩展:内部层通道数扩展为输入的 4倍。
- 作用:进行逐像素(Pixel-wise)的特征变换,类似 Dense Layer。
-
FCB (Fully Connected Block):
- 作用:处理全局信息,将张量映射为向量。
- 多维切片统计 (Slicing):不同于常规的全局平均池化 (GAP),FCB 为了保留更多空间分布信息,拼接了 4 种维度的统计量作为全连接层的输入:
- $c$:所有维度 $h, w$ 的平均(常规 GAP)。
- $c \times w$:仅在高度 $h$ 上平均。
- $c \times h$:仅在宽度 $w$ 上平均。
- $h \times w$:仅在通道 $c$ 上平均。
- 输入总维度:$c + cw + ch + hw$。
编码器
编码器的任务是将图像压缩为“DNA”。
-
Continuous Encoder:
- 使用堆叠的 CB3 和 CB1 提取图像特征。
- 通过 FCB 将空间特征图压缩为连续的潜在向量 (Latent Vector)。
-
DNA Encoder (Categorical Discretization):
- 目标是模拟生物 DNA 的离散特性 (A, C, G, T)。
- 结构:由 4 个连续的 CB1 (1D Conv) 组成。
- 输出:接 Softmax 层,输出 4 个类别的概率分布。
- 冗余设计:DNA 编码的维度设定为连续编码维度的 16倍。这种高冗余度使得模型对突变具有极强的鲁棒性(类似于生物 DNA 中非编码区或冗余备份的作用)。
解码器
解码器不直接生成像素,而是生成“规则”。
-
DNA Decoder:
- 与 DNA Encoder 对称,将离散的 DNA 序列映射回连续特征空间。
- 使用 1D 卷积 (CB1),独立处理每个“基因”位点。
-
Parameter Predictor (HyperNetwork):
- 输入:解码后的连续特征向量。
- 输出:一个巨大的扁平向量。
- Reshape:将输出向量重塑为 NCA 更新网络所需的卷积核权重 $\kappa(e^I)$。
- 动态卷积 (Dynamic Convolutions):这些生成的权重被用于定义 NCA 的更新函数:$Y^{I, n} = \sum_{m} \kappa(e^{I})_{mn} * X^{I, m}$。
NCA 执行模块
这是解码器的最后一步,负责实际的“生长”。
-
感知 (Perception):
- 使用固定的 Sobel 滤波器 提取梯度(同 Growing NCA)。
- 关键改进:Instance Normalization。
- 由于网络使用 ReLU(无上界),递归更新容易导致数值爆炸。
- 作者在 Sobel 滤波后引入 Instance Norm,强制规范化感知特征的分布,防止信号在时间步中指数级增长。这是 NCAM 训练稳定的关键。
-
更新 (Update):
- 使用由 Parameter Predictor 生成权重的动态卷积(本质是 1x1 卷积)。
- Leak Factor ($\theta_{LF}$):
- 这是一个可学习的标量参数,范围被限制在 $[10^{-3}, 10^3]$。
- 更新公式:$S^t = S^{t-1} + \text{LF} \cdot \Delta S$。
- 作用:类似于微分方程中的步长 $\Delta t$。较小的 LF 鼓励网络保留先前的信息,使生长过程更加平滑。
-
更新策略:
- 虽然训练时使用了随机更新 ($p=0.5$),但作者发现在推理时使用 同步更新 ($p=1.0$) 能显著降低误差(MSE 降低一个数量级),尤其是在生成精细纹理时。
实验结果
数据集与生成能力
- NotoColorEmoji (约 3000 张):能够精确重建绝大多数 Emoji。
- CIFAR-10 (50,000 张实景图):单一模型能够编码并重建 5 万张不同的自然图像。这证明了 NCAM 具有极大的容量,确实学习到了图像生成的通用流形。

基因工程
利用学习到的流形结构,作者展示了有趣的基因编辑操作:
- 均值基因移植:
- 计算一组具有共同特征图像(如“圆形物体”)的平均 DNA。
- 设定阈值,保留显著的基因片段。
- 将这些“圆形基因”注入到目标图像的 DNA 中。
- 结果:生成的图像保留了原始特征,同时融合了“圆形”特征。这证明潜空间具有良好的语义解耦性。

鲁棒性与损伤再生
虽然 NCAM 的权重是动态生成的,但只要 DNA 保持不变(即规则不变),生成的生物体依然继承了 NCA 的自修复能力。即使切除图像的一部分,它依然能再生回原样,因为这套动态生成的规则本身就是一个具有稳定吸引子的动力系统。
编码维度与冗余
实验表明,降低 Embedding 维度会导致图像质量下降,但增加维度(超过 512)并没有显著提升。 更有趣的是 DNA 编码的鲁棒性:由于设计了 16 倍的冗余度,即使在推理时对 DNA 引入 50% 的随机突变错误,模型生成的图像依然清晰可辨。这模拟了生物 DNA 中大量冗余信息对遗传稳定性的保护作用。
总结
NCAM 是 Neural Cellular Automata 的一次重要进化,它从“单一任务求解器”变成了“通用生成模型”。
- 架构创新:结合 Auto-Encoder、HyperNetwork 和 NCA,实现了“用一个网络生成无数个网络”。
- 生物学意义:成功模拟了从基因型(Genotype)到表型(Phenotype)的完整映射,包括转录调控和形态发生。
- 应用潜力:为设计可编程的、具有自组织和自修复能力的复杂系统提供了一种新的元学习框架。
评价
这篇论文的本质就是在原来的细胞自动机上面加了一个从图片到卷积参数的编码器,同时设计了一个不错的模型架构。说不上有多么的创新,但是可以学习一点他的方法和思想。